序列分析
获取最新的人的所有miRNA的ID号
代码的作用
加载了mirbase.rds这个文件,里面保存了人的所有miRNA的成熟体ID和miRNA名字。这样的话,我们就可以从TCGA中提取出全部的miRNA
准备文件
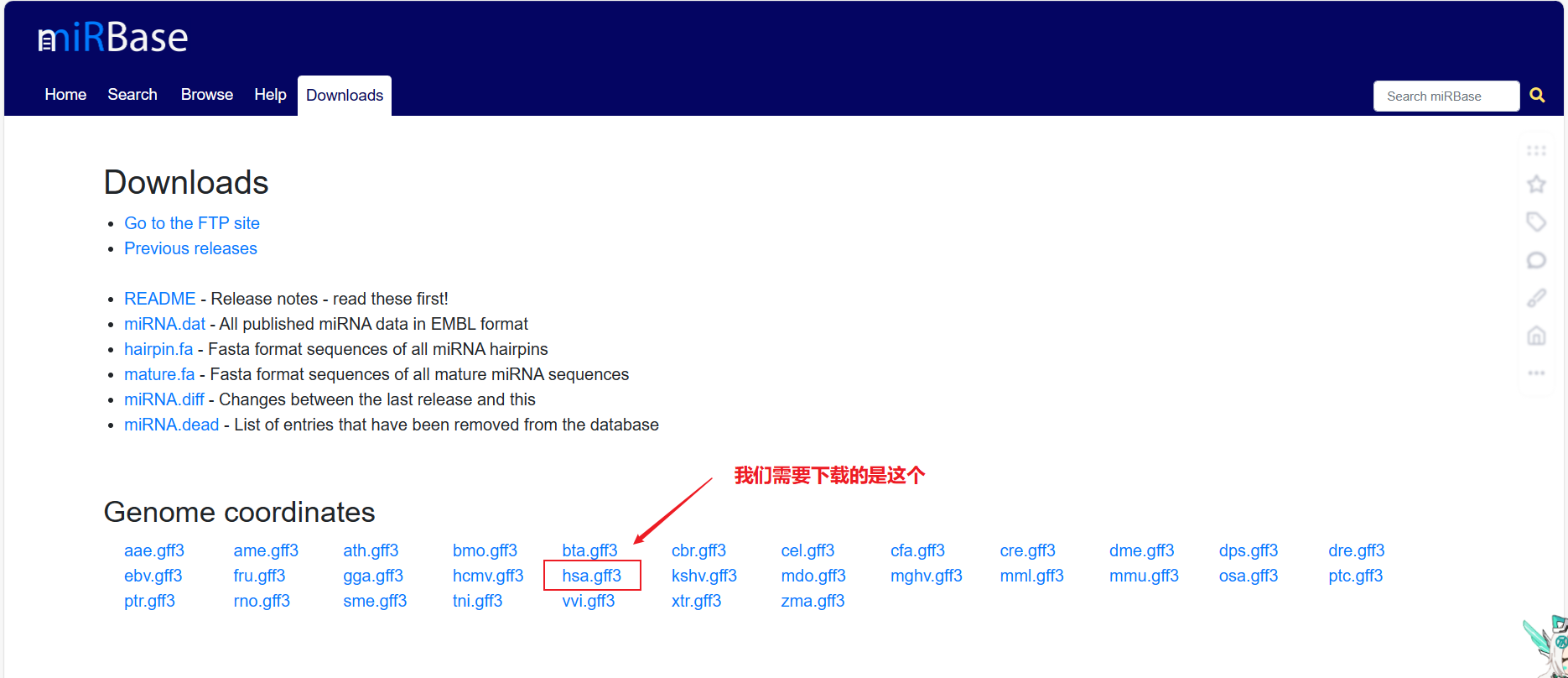
①需要到miRBase - Downloads这个网站下载我们需要的miRNA的信息
[!NOTE]- 具体下载详情
代码
[!NOTE]- 具体的代码
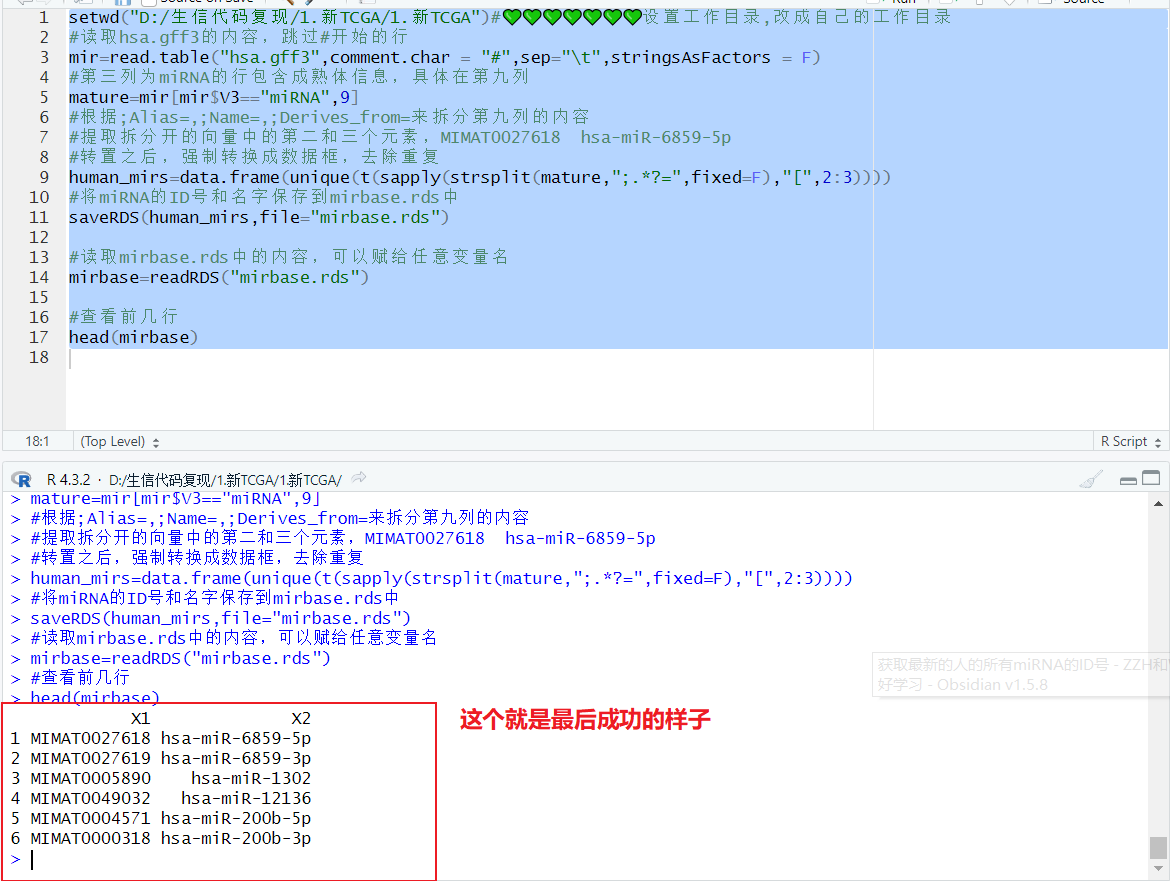
setwd("D:/生信代码复现/1.新TCGA/1.新TCGA")#💚💚💚💚💚💚💚设置工作目录,改成自己的工作目录 #读取hsa.gff3的内容,跳过#开始的行 mir=read.table("hsa.gff3",comment.char = "#",sep="\t",stringsAsFactors = F) #第三列为miRNA的行包含成熟体信息,具体在第九列 mature=mir[mir$V3=="miRNA",9] #根据;Alias=,;Name=,;Derives_from=来拆分第九列的内容 #提取拆分开的向量中的第二和三个元素,MIMAT0027618 hsa-miR-6859-5p #转置之后,强制转换成数据框,去除重复 human_mirs=data.frame(unique(t(sapply(strsplit(mature,";.*?=",fixed=F),"[",2:3)))) #将miRNA的ID号和名字保存到mirbase.rds中 saveRDS(human_mirs,file="mirbase.rds") #读取mirbase.rds中的内容,可以赋给任意变量名 mirbase=readRDS("mirbase.rds") #查看前几行 head(mirbase)
最终的效果
①在R语言中的效果
[!NOTE]- 在R中的效果
②在文件中的最终效果
[!NOTE]- 文件中的效果